※ DACON 링크
펭귄 몸무게 예측 경진대회 - DACON
좋아요는 1분 내에 한 번만 클릭 할 수 있습니다.
dacon.io
※ 변수 분석시 참고한 사이트
변수의 종류와 통계 기법
변수의 종류 수량적 특성에 의한 구분 질적 변수 (qualitative variables) 비서열 질적 변수 (unordered-qu...
blog.naver.com
해당 경진대회는 데이터 분석에 관심이 있는 사람들의 학습을 돕고, 실제 데이터 분석능력을 함양할 수 있게 해주는DACON에서 진행중인 온라인 기반 경진대회이다. Basic에 해당되어 분석할 때 참고할 수 있는 Baseline코드를 제공해주고 있으며, 해당코드를 활용하여 시작할 수 있다. 추가적으로 코드를 다른 사람들과 공유하면 순위권에 들지 않아도 상품을 제공해주고 있기에 다른 사람들도 다양한 분석코드를 올려주니 해당 내용도 참고하여 분석을 진행할 수 있어서 초심자들에게 좋은 대회이다. 데이터를 다운로드 받은 후 가장 먼저 데이터의 구조를 확인해보았다.
train.info()

# 결측값이 있는 행
for i in range(len((train.isna().sum(axis=1)>=1))):
if (train.isna().sum(axis=1)>=1)[i] == True:
print(i)

# na 값 삭제
train.dropna(inplace=True)
train.reset_index(drop=True, inplace=True)
결측치가 존재하는 것을 확인하여 결측치가 있는 행을 확인해보았다. 결측치는 총 9개였고, 결측값이 있는 행 자체는 5개 행이었다. 일단은 결측치를 제외시키고 분석을 진행하기로 결정하였으며, 결측치들은 전부 다 drop함수를 이용하여 제거해준 후 분석을 진행하였다. 그 후 변수의 형태가 object인 4개 요소들('Species', 'Island', 'Clutch Completion', 'Sex')의 데이터 분포형태를살펴보기 위해 matplotlib을 이용하여 시각화를 해보았다.
fig, axes = plt.subplots(2, 2, figsize = (12, 8), sharey = True)
ax = axes[0, 0]
sp_list = list(train.Species.unique())
sp_list_ch = []
for sp in sp_list:
sp_list_ch.append(sp.split('(')[0])
sp_sizes = []
for i in range(len(sp_list)):
globals()[f'sp_list_value{i}'] = len(train[train.Species == sp_list[i]].Species)
sp_sizes.append(globals()[f'sp_list_value{i}'])
# 파이차트 : 카테고리별 값의 상대적인 비교를 해야할 때 사용
labels = sp_list_ch
colors = ['yellowgreen', 'gold', 'lightskyblue']
explode = (0.1, 0, 0.1)
ax.pie(sp_sizes, explode = explode, labels = labels, colors = colors,
autopct = '%1.1f%%', shadow = True, startangle = 90)
ax.set_title('species')
ax = axes[0, 1]
land_list = list(train.Island.unique())
land_sizes = []
for i in range(len(sp_list)):
globals()[f'land_list_value{i}'] = len(train[train.Island == land_list[i]].Island)
land_sizes.append(globals()[f'land_list_value{i}'])
# 파이차트 : 카테고리별 값의 상대적인 비교를 해야할 때 사용
labels = list(train.Island.unique())
colors = ['red', 'yellow', 'blue']
explode = (0.1, 0.1, 0.1)
ax.pie(land_sizes, explode = explode, labels = labels, colors = colors,
autopct = '%1.1f%%', shadow = True, startangle = 90)
ax.set_title('Island')
ax = axes[1, 0]
CC_list = list(train['Clutch Completion'].unique())
CC_sizes = []
for i in range(len(CC_list)):
globals()[f'CC_list_value{i}'] = len(train[train['Clutch Completion'] == CC_list[i]]['Clutch Completion'])
CC_sizes.append(globals()[f'CC_list_value{i}'])
# 파이차트 : 카테고리별 값의 상대적인 비교를 해야할 때 사용
labels = list(train['Clutch Completion'].unique())
colors = ['blue', 'yellow']
explode = (0.1, 0.1)
ax.pie(CC_sizes, explode = explode, labels = labels, colors = colors,
autopct = '%1.1f%%', shadow = True, startangle = 90)
ax.set_title('Clutch Completion')
ax = axes[1, 1]
sex_list = list(train.Sex.unique())
sex_sizes = []
for i in range(len(sex_list)):
globals()[f'sex_list_value{i}'] = len(train[train.Sex == sex_list[i]].Sex)
sex_sizes.append(globals()[f'sex_list_value{i}'])
# 파이차트 : 카테고리별 값의 상대적인 비교를 해야할 때 사용
labels = list(train.Sex.unique())
colors = ['skyblue', 'yellowgreen']
explode = (0.1, 0.1)
ax.pie(sex_sizes, explode = explode, labels = labels, colors = colors,
autopct = '%1.1f%%', shadow = True, startangle = 90)
ax.set_title('Island')
plt.tight_layout()
plt.show()

시각화 결과를 확인한 후 Clutch Completion의 변수 분포가 Yes로 너무 치우쳐져 있어서 추후 변수처리를 할 때 유의하여 진행해야겠다고 생각하였으며, 추가적인 처리를 해주기 전에 일단 변수들간의 상관관계를 확인해보기로 결정하였다. 우선 간단하게나마 피어슨 상관계수를 확인해보기로 결정하였고, 코드를 작성하기 전 명목변수들의 인코딩을 진행해주었다.
# 데이터 분석을 위한 원-핫 인코딩
sp_list = list(train.Species.unique())
sp_index = [x for x in range(len(sp_list))]
sp_dict = dict(zip(sp_list, sp_index))
train.Species = train.Species.apply(lambda x: sp_dict[x])
land_list = list(train.Island.unique())
land_index = [x for x in range(len(land_list))]
land_dict = dict(zip(land_list, land_index))
train.Island = train.Island.apply(lambda x: land_dict[x])
CC_list = list(train['Clutch Completion'].unique())
CC_index = [x for x in range(len(CC_list))]
CC_dict = dict(zip(CC_list, CC_index))
train['Clutch Completion'] = train['Clutch Completion'].apply(lambda x: CC_dict[x])
sex_list = list(train.Sex.unique())
sex_index = [x for x in range(len(sex_list))]
sex_dict = dict(zip(sex_list, sex_index))
train.Sex = train.Sex.apply(lambda x: sex_dict[x])
train.drop(['id'], axis=1, inplace=True)
train.dtypes
인코딩 후에는 상관계수를 확인하여 상관계수가 높은 순으로 정렬하여 확인해보았고, 전체적인 상관계수를 확인하기 위해 시각화를 하여 확인을 해보았다.
# 상관계수 확인(절댓값)
abs(train.corr()['Body Mass (g)'].drop(['Body Mass (g)'])).sort_values(ascending=False)

colormap = plt.cm.RdBu
plt.figure(figsize = (12, 12))
plt.title('corr score')
sns.heatmap(train.corr(), linewidth = 0.1, vmax = 1.0, square = True, \
cmap = colormap, linecolor = 'white', annot = True, annot_kws = {'size' : 10})

확인결과, 대부분의 변수들이 상관관계가 있는 것으로 확인되었으나, 독립변수들간의 상관성도 높은 것들이 있어 추가적인 분석을 통해 변수를 선정할 필요가 있다고 생각하였다. 그래서 명목변수들과 종속변수(몸무게)와의 상관성을 확인해보기 위해 크루스칼-왈리스 검정을 실시하고, 독립변수들의 가중치가 중복적으로 적용되는 것을 확인하고 제거하기 위해 명목변수들간 카이스퀘어 검정을 실시하기로 하였다. 추가적으로 연속변수들을 stats패키지를 활용하여 p-value 확인 후 제거할 변수들을 결정하기로 하였다.
# 크루스칼 왈리스 검정 실시 # 등분산성 및 정규성 검정은 미실시
from scipy import stats
import statsmodels.api as sm
from statsmodels.formula.api import ols
# p-value가 0.05이하이므로 귀무가설(관계가 없다)기각 - Species는 관계가 있음
print('Species:', stats.kruskal(train[train.Species == 0]['Body Mass (g)'],\
train[train.Species == 1]['Body Mass (g)'], train[train.Species == 2]['Body Mass (g)']))
# p-value가 0.05이하이므로 귀무가설(관계가 없다)기각 - Island는 관계가 있음
print('Island:', stats.kruskal(train[train.Island == 0]['Body Mass (g)'],\
train[train.Island == 1]['Body Mass (g)'], train[train.Island == 2]['Body Mass (g)']))
# p-value가 0.05이하이므로 귀무가설(관계가 없다)기각 - Sex는 관계가 있음
print('Sex:', stats.kruskal(train[train.Sex == 0]['Body Mass (g)'],\
train[train.Sex == 1]['Body Mass (g)']))
# p-value가 0.05이상이므로 귀무가설(관계가 없다)기각불가 - Clutch Completion는 관계가 없음
print('Clutch Completion:', stats.kruskal(train[train['Clutch Completion'] == 0]['Body Mass (g)'],\
train[train['Clutch Completion'] == 1]['Body Mass (g)']))

from statsmodels.formula.api import ols
from statsmodels.api import OLS, add_constant
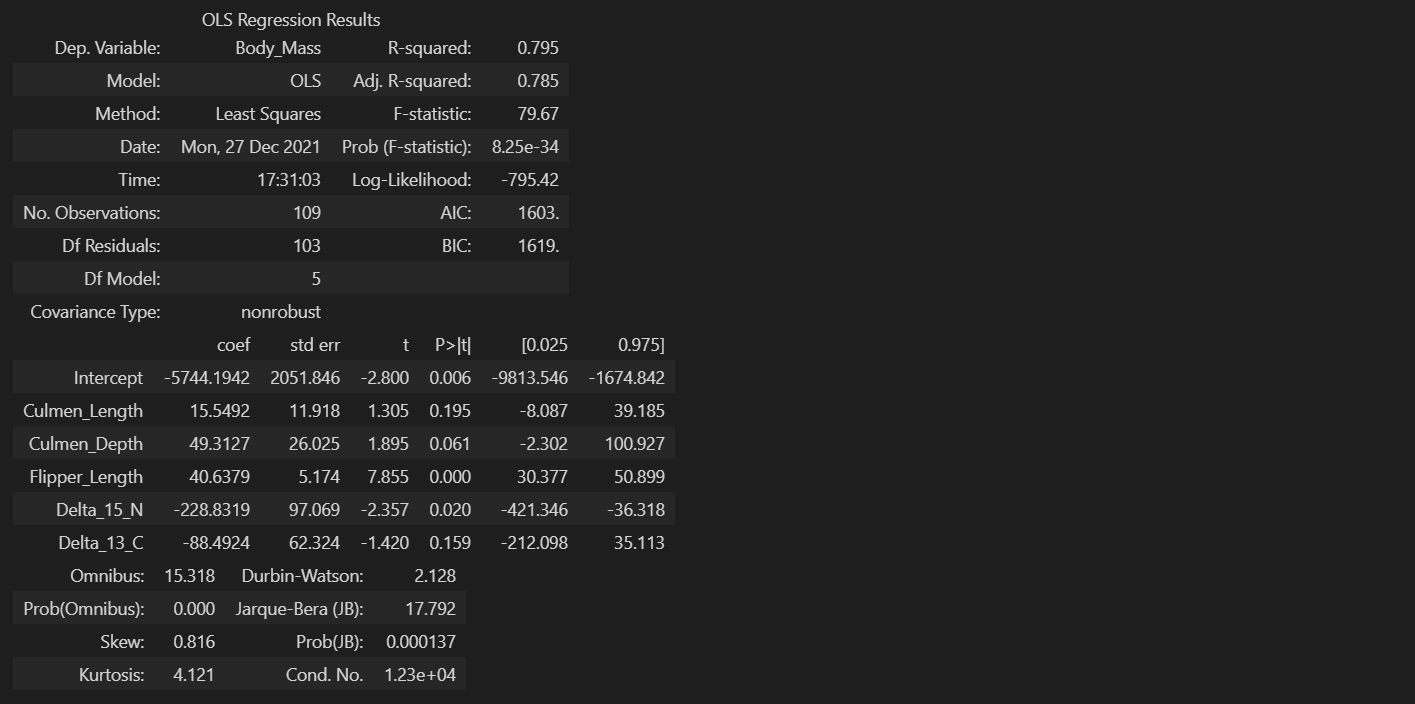
x_list = ['Culmen_Length', 'Culmen_Depth', 'Flipper_Length', 'Delta_15_N', 'Delta_13_C']
formula = 'Body_Mass~' + '+'.join(x_list)
model = OLS.from_formula(formula, data = train[x_list + ['Body_Mass']])
result = model.fit()
# print(result.params)
result.summary()
# ols = Ordinary least square(최소 자승법 : 잔차제곱합을 최소화하는 가중치 벡터를 구하는 방법)
# Df Model : 독립변수의 개수 // Df(자유도) residuals : 전체 표본개수에서 독립변수, 종속변수의 개수를 뺀 것
# intercept coef: 절편 // 독립변수 coef : 기울기
# Dep.Variable : Dependent variable = 종속변수 // Model : 모델링 방법
# R squared : 결정계수 - 전체 데이터중 해당 모델이 설명할 수 있는 데이터의 비율 = 설명력 / 값이 크면 예측이 정확(0~1)
# F-statistics : F 통계량 - 값이 크면 선형이 잘 안그려짐(0 ~ inf)
# Prob : F 통계량에 해당하는 P-value
# p-value가 0.05이상인 변수들 제거 - Delta_13_C, Culmen_Length, Culmen_Depth
# dfs를 이용한 카이스퀘어를 실시할 변수 조합 작성
col_name = ['Species', 'Island', 'Clutch Completion', 'Sex']
check_list = [False] * len(col_name)
com_list = []
arr = []
def dfs(start):
if len(arr) == 2:
com_list.append(arr[:])
return
for i in range(start, len(col_name)):
if check_list[i]:
continue
check_list[i] = True
arr.append(col_name[i])
dfs(i)
arr.pop()
check_list[i] = False
dfs(0)
# 명목변수들간 상관계수 비교
from scipy.stats import chi2_contingency
import pandas as pd
for i in range(len(com_list)):
globals()[f'ct{i}'] = pd.crosstab(train[com_list[i][0]], train[com_list[i][1]], margins = False) # margin을 True를 넣으면 빈도수의 합까지 출력해줌
globals()[f'result{i}'] = chi2_contingency(observed = globals()[f'ct{i}'], correction=False)
print(f"* {com_list[i][0]} & {com_list[i][1]}")
print("chi - statistic:", globals()[f'result{i}'][0])
print("p-value:", globals()[f'result{i}'][1]) # p-value의 값이 0.05보다 낮으면 두 변수는 독립이 아님.(귀무가설 기각) - 연관이 있음.
if globals()[f'result{i}'][1] > 0.05:
print(f'{com_list[i][0]} 와 {com_list[i][1]}은 서로 연관성이 없음')
else:
print(f'{com_list[i][0]} 와 {com_list[i][1]}은 서로 연관성이 있음')
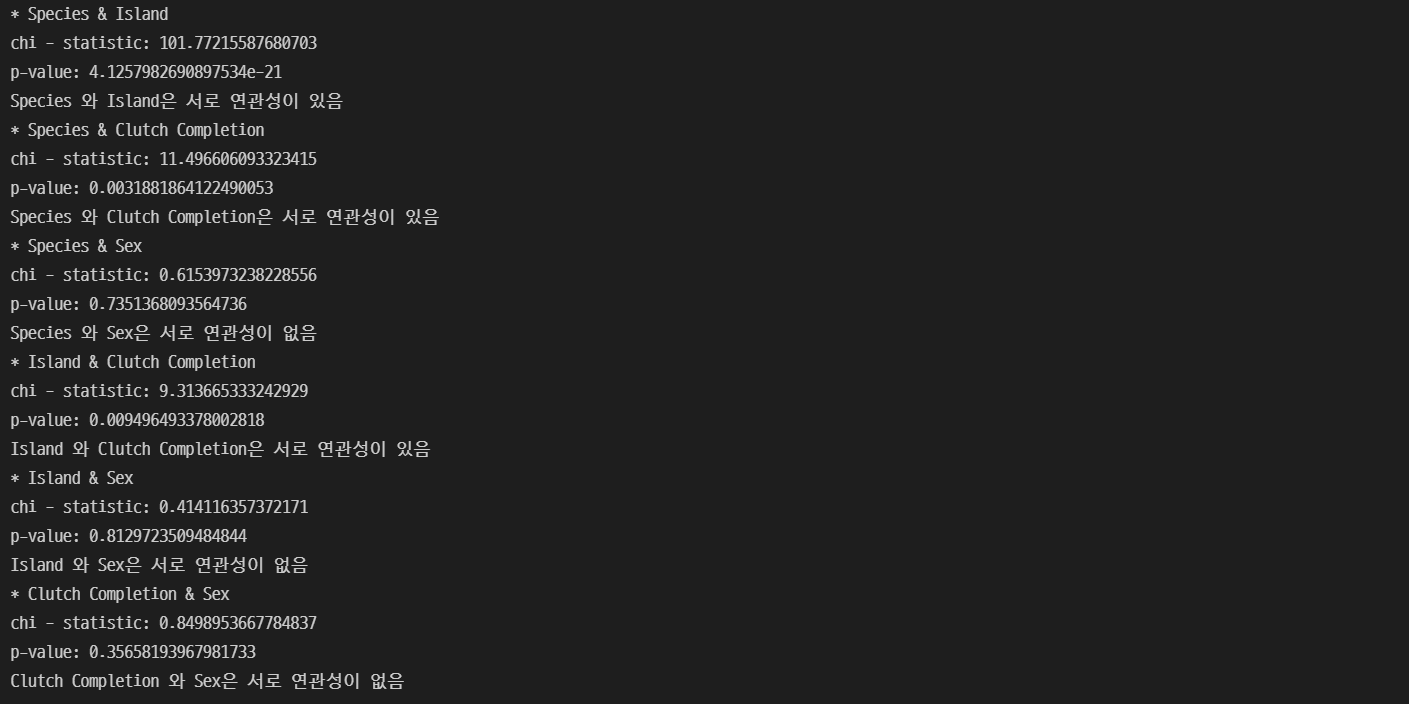
분석결과, Clutch Completion과 몸무게와의 연관성은 없다고 확인하여 해당 변수는 분석에서 제거하기로 결정하였고, 독립변수들 중 연속변수들 중 Culmen_Length, Culmen_Depth, Delta_13_C는 p-value가 0.05이상로 나타난 것으로 확인하여 제거하고 분석을 하기로 결정하였다. 명목변수들 중 Species와 Island의 연관성이 높다고 판단하여, 둘 중 종속변수인 몸무게와 연관성이 더 높은 Species만 사용하여 분석을 하기로 결정하였다. 최종적으로 분석에는 Species, Sex, Flipper_Length, Delta_15_N 4개 변수만 사용하기로 결정하였고, 그 후 테스트 데이터의 구조를 확인해보았다.
test.isnull().sum()

null_list = []
for i in range(len((test.isna().sum(axis=1)>=1))):
if (test.isna().sum(axis=1)>=1)[i] == True:
null_list.append(i)
test.iloc[null_list, :]
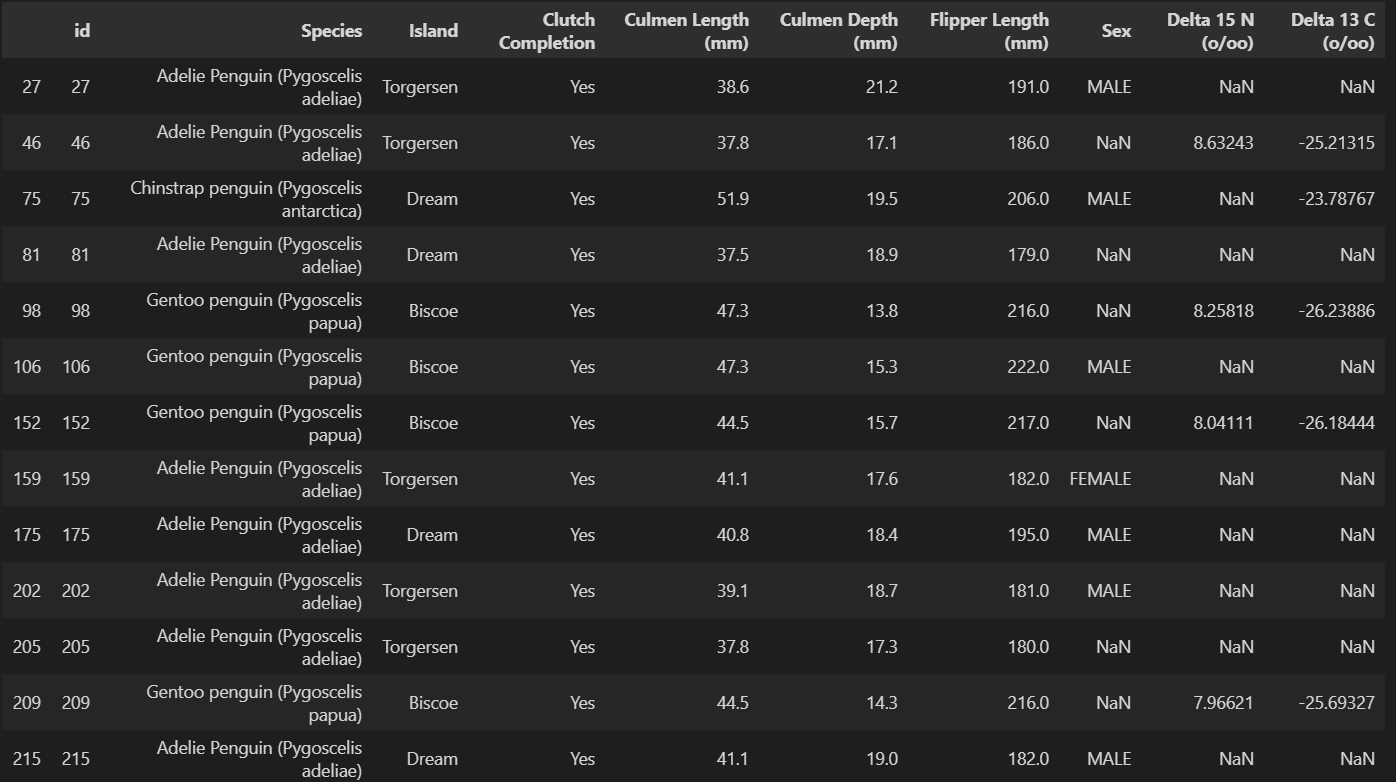
결측값은 총 23개로 Sex, Delta_15_N, Delta_13_C에만 존재하는 것으로 확인하였다. 예측에 사용할 Sex와 Delta_15_N에 있는 결측값만 처리를 해주기로 결정하였고, 일단은 간단하게만 처리해주기로 결정하였다. Sex결측값은 Flipper_Length의 성별 평균치를 확인해보앗는데, 수컷이 암컷에 비해 길이가 더 긴것으로 확인하여, Flipper_Length의 길이의 평균보다 길면 수컷으로, 짧으면 암컷으로 처리를 해주었다. Delta_15_N은 훈련데이터의 평균값을 구해 해당 값으로 처리하였다. 예측모델은 가장 기본적인 sklearn의LinearRegression모델을 사용하기로 하였고, RMSE 함수는 DACON에서 제공하는 코드를 활용하여 결과값을 확인해보았다.
# Linear Regression 로드 및 확인
from sklearn.linear_model import LinearRegression
x_list = ['Flipper_Length', 'Species', 'Sex', 'Delta_15_N']
lm = LinearRegression()
lm.fit(train[x_list], train.Body_Mass)
print(lm.coef_)
lm.predict(train[x_list]) - train.Body_Mass
# RMSE 함수
def RMSE(true, pred):
score = np.sqrt(np.mean(np.square(true-pred)))
return score
RMSE(lm.predict(train[x_list]), train.Body_Mass)
P.S 개발자가 되기위해 공부중입니다. 잘못된 부분을 댓글로 남겨주시면 학습하는데 큰 도움이 될 거 같습니다.
세부코드가 궁금하신 분들은 아래 GitHub를 참고해주시기 바랍니다.
※ Github
GitHub - Jeong-Beom/TIL: 교육받은 내용을 기록하기 위한 레파지토리입니다.
교육받은 내용을 기록하기 위한 레파지토리입니다. Contribute to Jeong-Beom/TIL development by creating an account on GitHub.
github.com
'IT & 데이터 사이언스 > 데이터 분석 실습' 카테고리의 다른 글
| [DACON] 영화 리뷰 감성분석 경진대회(Basic) (0) | 2022.01.22 |
|---|---|
| [DACON] 심장질환예측 경진대회(Basic)(3) (0) | 2021.12.23 |
| [DACON] 심장질환예측 경진대회(Basic)(2) (0) | 2021.12.18 |
| [DACON] 심장질환예측 경진대회(Basic)(1) (0) | 2021.12.18 |




댓글